dataforgoodfr / Batch_5_transplant
Labels
Projects that are alternatives of or similar to Batch 5 transplant
batch_5_transplant
Présentation
La transplantation pulmonaire donne une seconde chance à des patients, souvent jeunes, condamnés par une insuffisance respiratoire chronique.
L'objectif du projet, en collaboration avec des médecins spécialistes de l’hôpital Foch (1° centre de transplantation pulmonaire français) est d'aider à la décision de retirer l'assistance respiratoire en fin d’opération.
Le sevrage de l’assistance respiratoire en fin d’opération, marqueur du bon déroulement de l’opération permet de diminuer le risque nosocomial et de mettre le patient dans une situation active le plus rapidement possible.
Une opération de transplantation se fait en plusieurs phases:
- Anesthésie générale avec mise en condition du patient (sédation,support hémodynamique, ventilation assistée voire ECMO (extra corporeal Membran oxygenation)
- Explantation du premier poumon et implantation du premier nouveau poumon
- Explantation du second poumon et implantation du second nouveau poumon
- Réveil et si possible sevrage
Le succès de la chirurgie dépend de facteurs liés au receveur, au greffon et à différents évènements per opératoires. C’est l’analyse de ces différents paramètres qui pourrait permettre d’optimiser la prise en charge et la décision du sevrage ventilatoire en fin d’intervention
Organisation du Repository
├── data
│ ├── clean <- The final, canonical data sets for modeling. (in .gitignore)
│ └── raw <- The original, immutable data dump.
│
├── docs <- Documentation
│
├── models <- Trained and serialized models, model predictions,
│ or model summaries. (in .gitignore)
├── notebooks <- Jupyter notebooks.
│
├── reports <- Generated analysis as HTML, PDF, LaTeX, etc.
│ └── figures <- Generated graphics and figures to be used in reporting
│
├── transplant <- Source code for use in this project.
│ ├── data <- Scripts to generate and get data (dataset.py class for instance)
│ │
│ ├── features <- Scripts to turn raw data into features for modeling
│ │
│ ├── models <- Scripts to train models and then use trained models to make
│ │ predictions
│ │
│ ├── tests <- Unittests for this project
│ │
│ └── visualization <- Scripts to create exploratory/results oriented visualizations
│
├── README.md <- The top-level README for developers using this project.
├── requirements.txt <- The requirements file for reproducing the analysis environment
└── setup.py <- Make this project pip installable with `pip install -e`
Dataset
L'hôpital Foch met à disposition un historique de 412 patients ayant reçu une transplantation pulmonaire depuis Janvier 2012. Le dataset contient:
- Variables caractérisant le receveur avant l’opération
- Variables caractérisant le greffon avant implantation
- Séries temporelles extraites des instruments de mesure du bloc opératoire.
- Marqueurs temporels collectés manuellement lors de la transplantation.
Setup
Il faut tout d'abord cloner le repo dans un dossier avec :
git clone [email protected]:dataforgoodfr/batch_5_transplant.git
Ensuite, il y a deux methodes possibles pour setup le projet :
1ère methode : avec pip install
Placez vous dans le dossier racine du repo (batch_5_transplant/), et exécuter la commande suivante pour installer le package transplant :
pip install -e .
2ème méthode : avec le PYTHONPATH
Ajoutez le repo au sein de votre PYTHONPATH.
Pour Mac :
Voici un exemple pour Mac, où le repo a été cloné au sein du dossier ~/Documents:
open ~/.bashrc
export PYTHONPATH="/Users/username/Documents/batch_5_transplant:$PYTHONPATH"
Pour Windows :
Rendez-vous dans 'Paramètres système avancés' puis dans “Variables d’environnement…”. Il faut créer une nouvelle variable système ici :
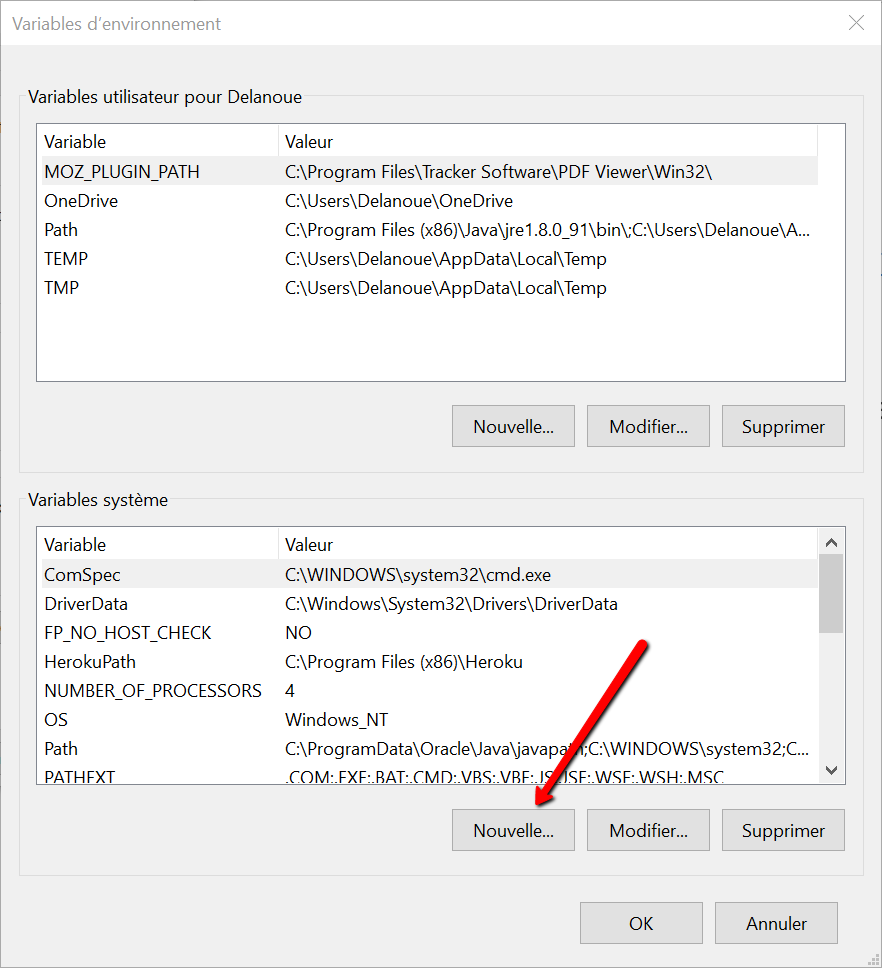
Et remplir la fenêtre de la manière suivante en remplaçant le texte dans ‘Valeur de variable’ par le chemin vers votre propre dossier ‘batch_5_transplant’ :
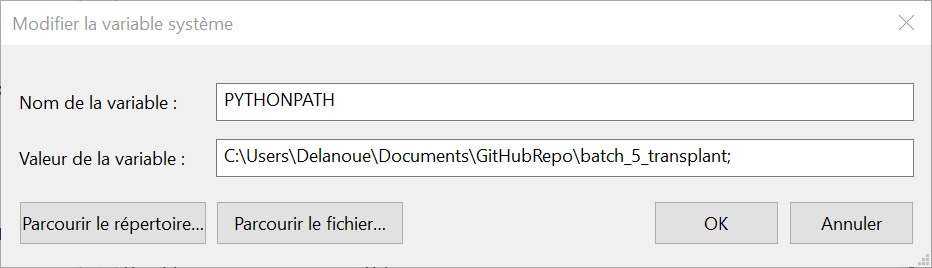
Et voilà ! N’oubliez pas de rouvrir votre jupyter. (Pour vérifier si la nouvelle variable est bien active écrivez ‘ ! set ‘ dans une cellule jupyter pour visualiser toutes les variables systèmes actives.
Pour en savoir plus sur les environnements et les PYTHONPATH voir ce lien pour MAC ou ce lien pour Windows
Vérifier l'installation
Vous pouvez desormais importer depuis un notebook les classes utiles au traitement de données avec la commande:
from transplant.data.dataset import Dataset
FAQ
Nous maintenons une foire aux questions au sein de ce document partagé.
Arborescence
Vous retrouverez plusieurs dossiers dans ce git:
-
data: vous retrouverez dans ce dossier l'ensembles des datasets. -
documentation: dossier contenant tous les fichiers de documentation. -
scripts: dossier contenant les scripts de cleaning et création des tables de dimensions et faits. -
etudes: pour chaque étude effectuée merci de mettre vos notebooks dans ce dossier. -
production: classes et fonctions permettant de standardiser l'import des données, training set et evaluation des modèles.
Images
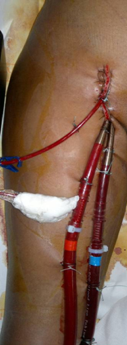
Image 1 - eCMO
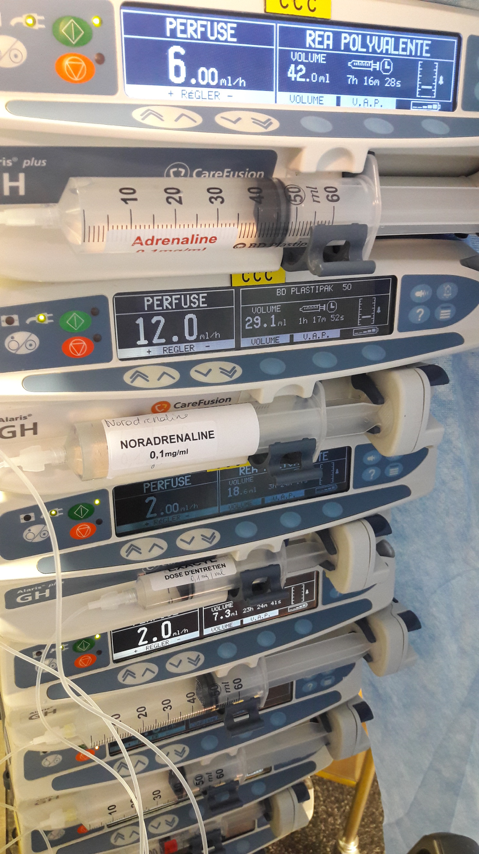
Image 2 - de nombreux traitements sont administrés pendant la chirurgie.
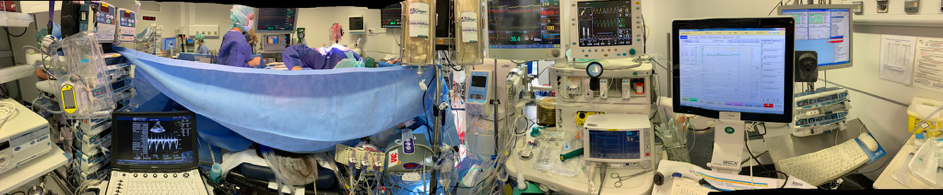
Image 3 - outils de monitoring continu pendant la chirurgie.
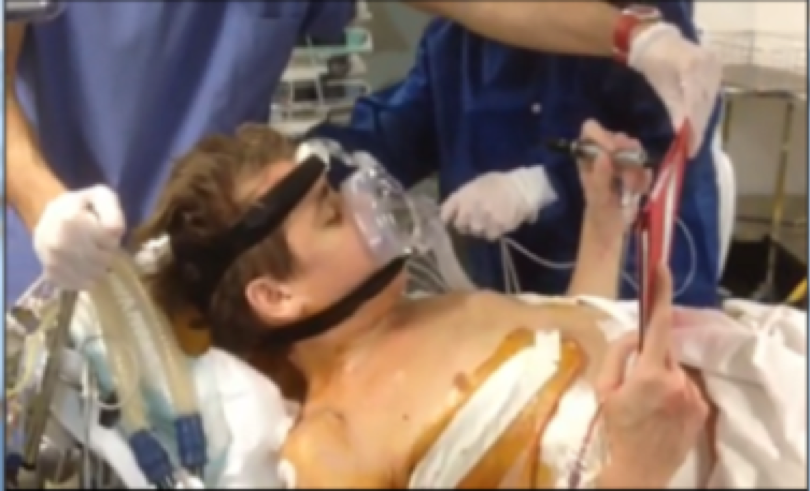
Image 4 - patient extubé en salle d’opération écrivant un message