Análise da COVID19 no Brasil
Análise do surto de COVID19 no Brasil empregando-se principalmente modelos epidemiológicos e de processos hospitalares.
ADVERTÊNCIA: os modelos e números aqui apresentados não são afirmações formais médicas sobre o progresso da doença, mas apenas exercícios que demonstram técnicas de modelagem e cenários hipotéticos de aplicação.
Introdução
A recente pandemia de COVID-19 vem motivando uma série de iniciativas ao redor do mundo para entender sua dinâmica e facilitar o trabalho de profissionais e gestores de saúde, no âmbito tanto público quanto privado. Aqui apresentamos uma contribuição por meio da implementação de análises, modelos epidemiológicos, estudos de processos de saúde e outros exercícios variados na linguagem Python, com o objetivo de permitir a cientistas de dados, principalmente no Brasil, a terem um ponto de partida a partir do qual conduzirem seus próprios estudos. Na medida do possível, buscaremos também exercitar esses modelos para produzir alguns resultados numéricos sugestivos, porém por enquanto é preciso advertir que esses números não são afirmações formais sobre o progresso da doença, mas apenas exercício que demosntram o uso dos modelos propostos. Pequenas mudanças nos parâmetros dos modelos podem levar a vastas mudanças nos resultados.
O repositório está organizado nas seguintes pastas:
data/: Dados históricos da COVID19, usado para estimação de parâmetros dos modelos.data/large/: Dados históricos grandes demais para serem versionados. Estes dados podem ser baixados usando o script de download fornecido (ver abaixo).data/preprocessed/: Dados pré-processados para conveniência de algumas análises. São criados por um notebook específico (ver abaixo).
notebooks/: Notebooks Jupyter com as análises e modelos.results/: Resultados de análises e modelos, para reuso em outros contextos.results/eda/: Imagens e CSVs resultantes das análises exploratórias de dados.results/notebooks/: Com o uso da biblioteca papermill, os próprios notebooks podem ser customizados resultarem em novas versões, que são então nesta pasta.results/notebooks/Brazil: Resultados específicos para estados e municípios brasileiros, valendo-se dos dados disponibilizados pelo Brasil.io
Destacamos ainda dois mecanismos de dados úteis de modo geral:
download_data.sh: Script que baixa os dados necessários para as diversas análises e modelos. Para ambientes Unix / Linux.notebooks/data_preprocessing.ipynb: notebook que pré-processa dados e os coloca em formato mais conveniente para algumas análises.
Modelos Epidemiológicos
Estão implementados alguns modelos epidemiológicos clássicos, a saber:
- SIR (Susceptible-Infectious-Recovered): ver notebook
epidemic_model_sir.ipynb. Este é um modelo simples que usamos apenas para demonstrar as técnicas básicas envolvidas. - SEIR (Susceptible-Exposed-Infectious-Recovered): ver notebook
epidemic_model_seir.ipynb. Este modelo é uma sofisticação do SIR. Ademais, focamos melhorias e análises nele.
Também fornecemos outras formas de modelagem:
- Aprendizado de Máquina clássico: ver notebook
epidemic_model_ml.ipynb. Aqui exploramos a aplicação de alguns algoritmos clássicos de Aprendizado de Máquina diretamente sobre os dados epidemiológicos. Não acreditamos que essa abordagem seja muito eficaz, posto que despreza conhecimento a priori sobre a dinâmica do processo sendo aprendido, porém é fornecida como base de comparação, e eventualmente como ponto de partida para experimentos mais sofisticados.
Esses notebooks podem ser baixados por interessados e customizados de diversos modos. No próprio texto de cada um apresentamos algumas idéias e exercícios, que podem servir de base para estudos e modelos mais complexos.
Ademais, fornecemos um notebook central, models.ipynb, por meio do qual os mesmos modelos podem ser re-executados com diversas variações de parâmetros, mediante o uso da biblioteca papermill.
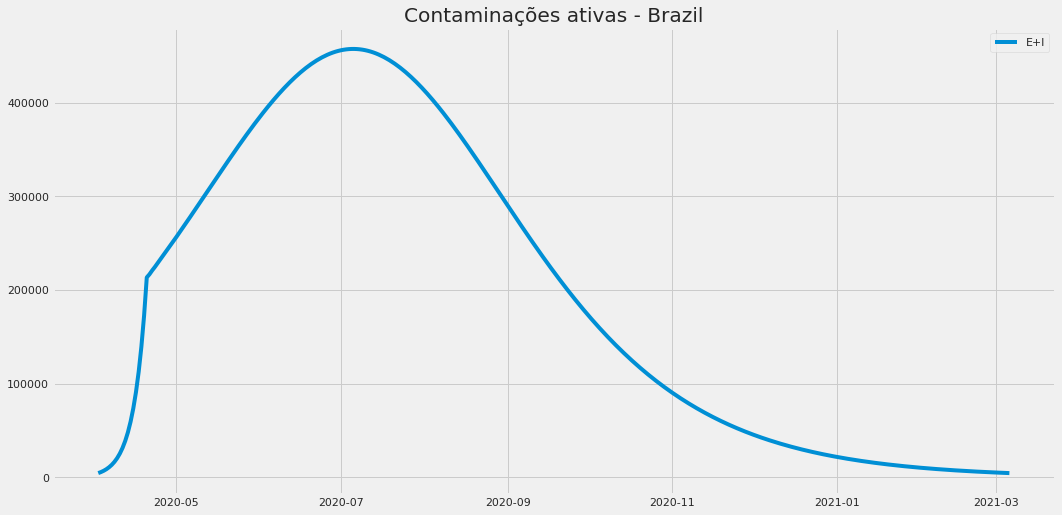 Exemplo de saída em um dos exercícios com o modelo SEIR. Os números são meramente ilustrativos.
Exemplo de saída em um dos exercícios com o modelo SEIR. Os números são meramente ilustrativos.
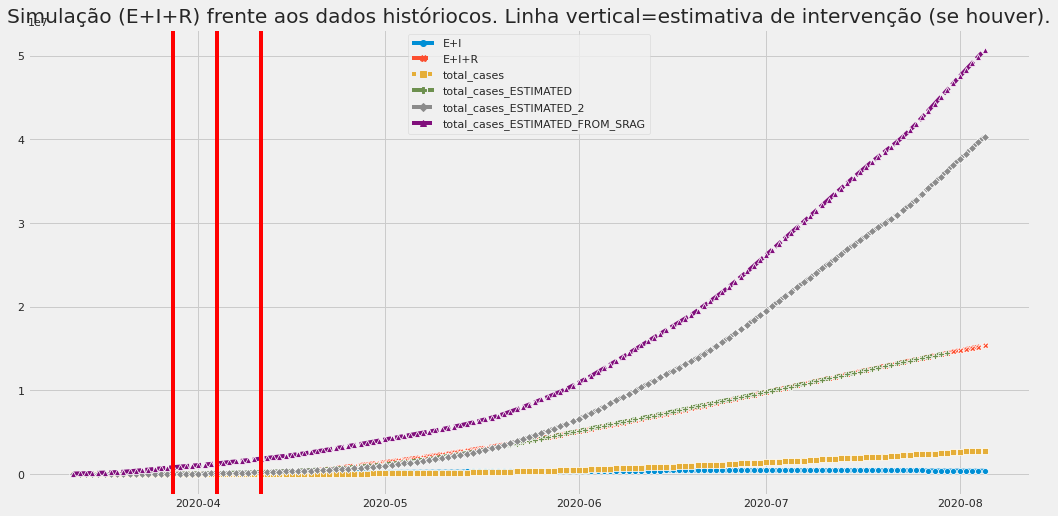 Exemplo de dados de simulação ajustados aos dados observados em um dos exercícios com o modelo SEIR. Os números da previsão são meramente ilustrativos.
Exemplo de dados de simulação ajustados aos dados observados em um dos exercícios com o modelo SEIR. Os números da previsão são meramente ilustrativos.
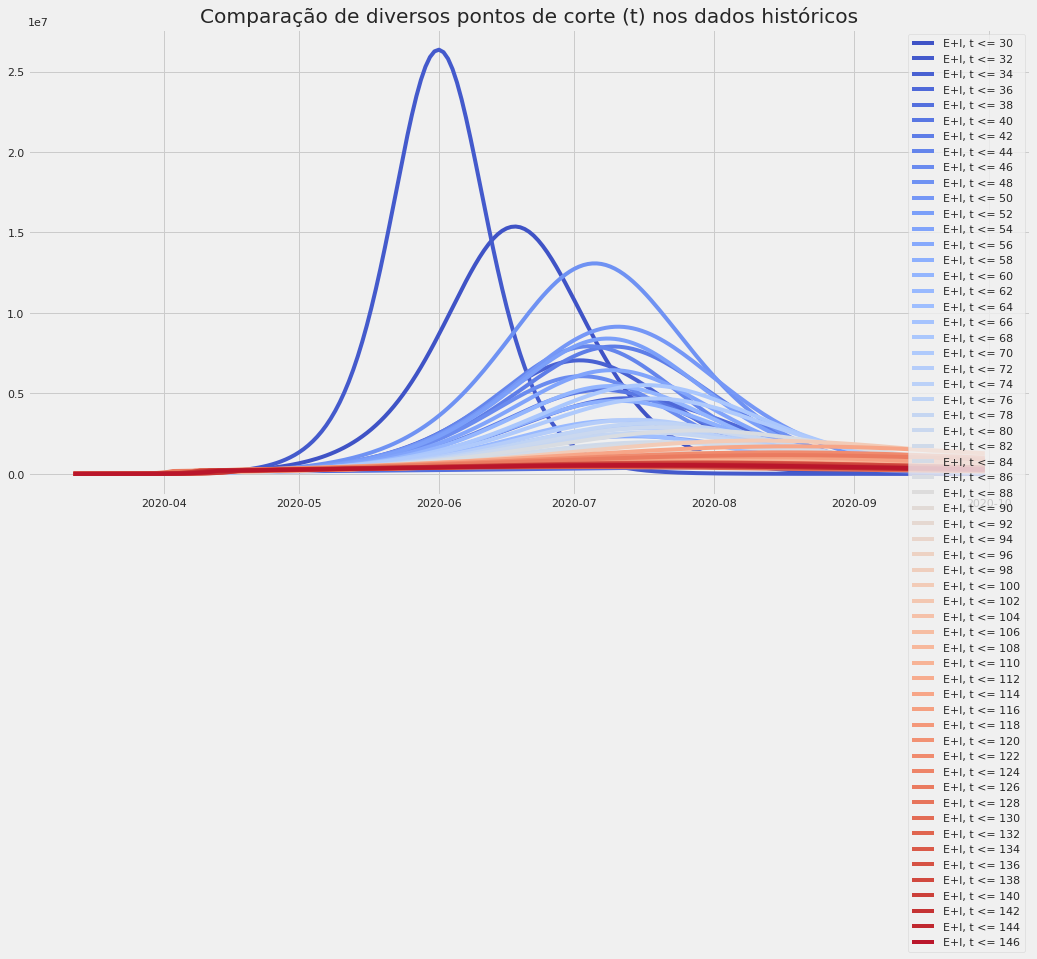 Exemplo de diversos ajustes do modelo SEIR, considerando partes dos dados para o Brasil. Note como há grande sensibilidade dos resultados, por isso enfatizamos que os números da previsão são meramente ilustrativos.
Exemplo de diversos ajustes do modelo SEIR, considerando partes dos dados para o Brasil. Note como há grande sensibilidade dos resultados, por isso enfatizamos que os números da previsão são meramente ilustrativos.
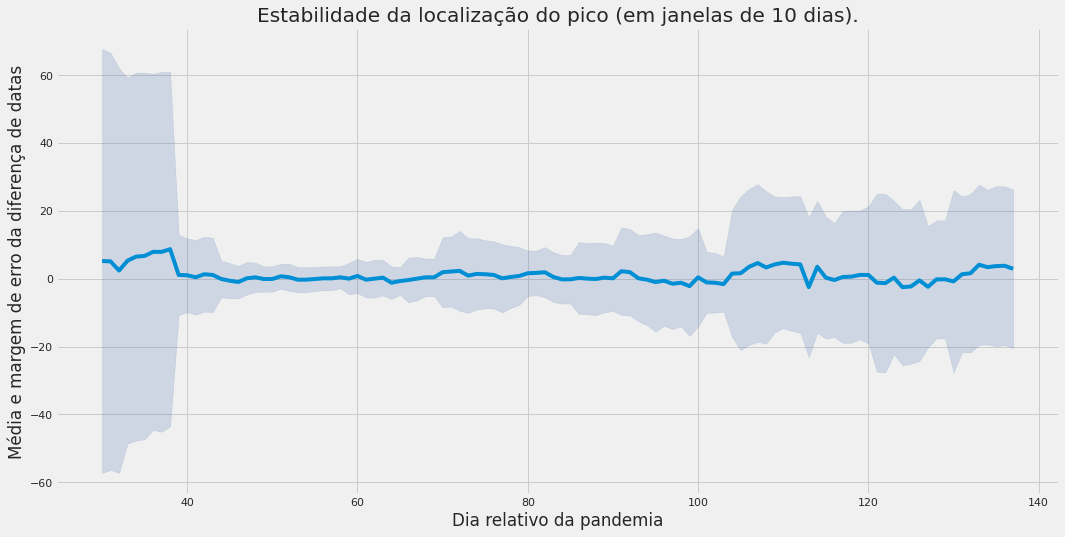 Exemplo de análise de sensibilidade do modelo SEIR, considerando partes dos dados para o Brasil. Note que a posição do pico varia muito no início, passa por um período de pouca variabilidade, e passa ficar novamente instável. Essa análise pode ser um modo interessante de quantificar a confiabilidade das previsões sendo feitas.
Exemplo de análise de sensibilidade do modelo SEIR, considerando partes dos dados para o Brasil. Note que a posição do pico varia muito no início, passa por um período de pouca variabilidade, e passa ficar novamente instável. Essa análise pode ser um modo interessante de quantificar a confiabilidade das previsões sendo feitas.
Aplicação Interativa
Também fornecemos uma aplicação interativa para simular o modelo SEIR, fazendo-se uso da biblioteca streamlit. Com isso, pode-se facilmente explorar os dados de diversos países, e aplicar-se o modelo SEIR a cada um deles com os parâmetros desejados. Para executá-la, basta garantir que a biblioteca e demais dependência estejam instaladas e então executar:
streamlit run src/app_interactive_seir/run.py
Ou então, mais convenientemente, o script seguinte (em ambientes Unix / Linux):
run_seir_app.sh
Se necessário, as dependências Python podem ser todas instaladas com o seguinte comando:
pip install -r requirements.txt
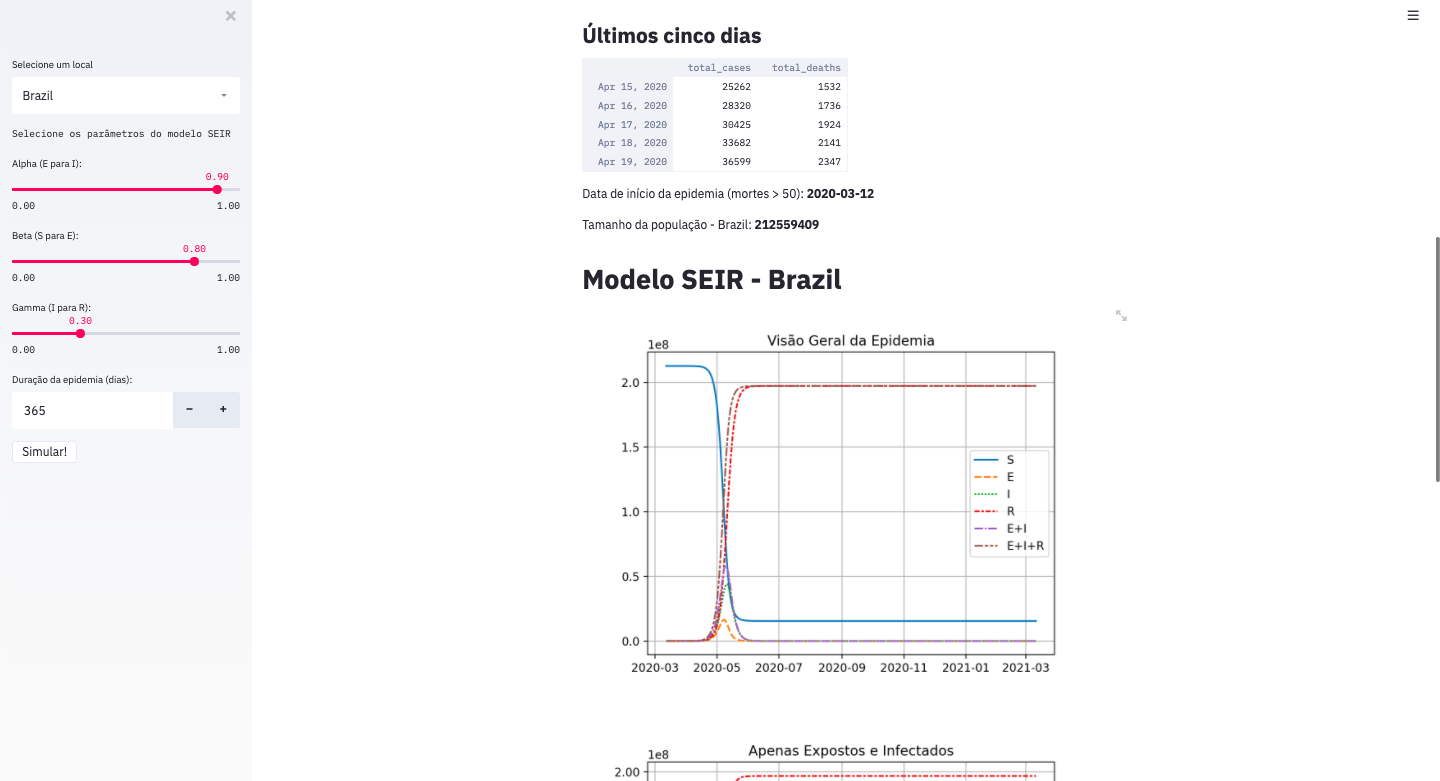 Exemplo de tela da aplicação interativa executando modelo SEIR. Os números são meramente ilustrativos.
Exemplo de tela da aplicação interativa executando modelo SEIR. Os números são meramente ilustrativos.
Modelos de Processos Hospitalares
Além da epidemia em si, é útil compreender como o sistema de saúde se comporta frente aos números projetados de infecções. Para tanto, também implementamos um modelo de processos hospitalares no notebook hospitalization_process.ipynb.
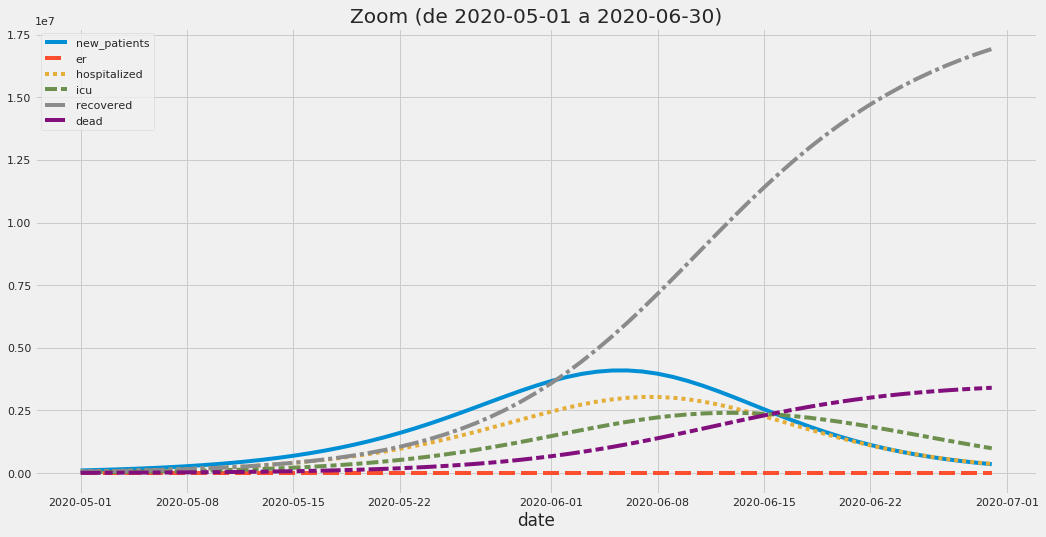 Exemplo de saída em um dos exercícios com o modelo de processos hospitalares. Os números são meramente ilustrativos.
Exemplo de saída em um dos exercícios com o modelo de processos hospitalares. Os números são meramente ilustrativos.
Análises de Dados Exploratórias
Fazemos algumas análises em dados disponíveis publicamente, notoriamente os do Our World in Data. Temos os seguintes notebooks disponíveis:
eda.ipynb: Centraliza a execução de outros notebooks com parâmetros customizados, mediante o uso da biblioteca papermill. Assim, uma mesma análise pode ser re-executada com diversas variações de parâmetros. Os resultados dessas análises customizadas são também notebooks, que são colocados emresults/notebooks/.eda_international.ipynb: Análises comparativas variadas do progresso da detecção de casos e óbitos ao redor do mundo. Esta análise gera diversos gráficos e dados comparativos, que são armazenados na pastaresults/para conveniência.eda_srag.ipynb: Análises dos casos de Síndrome Respiratória Aguda Grave (SRAG) no Brasil, fazendo-se uso dos dados da Fiocruz. Permite estimar casos não reportados de COVID-19, caso tenham sido registrados ao menos como SRAG.
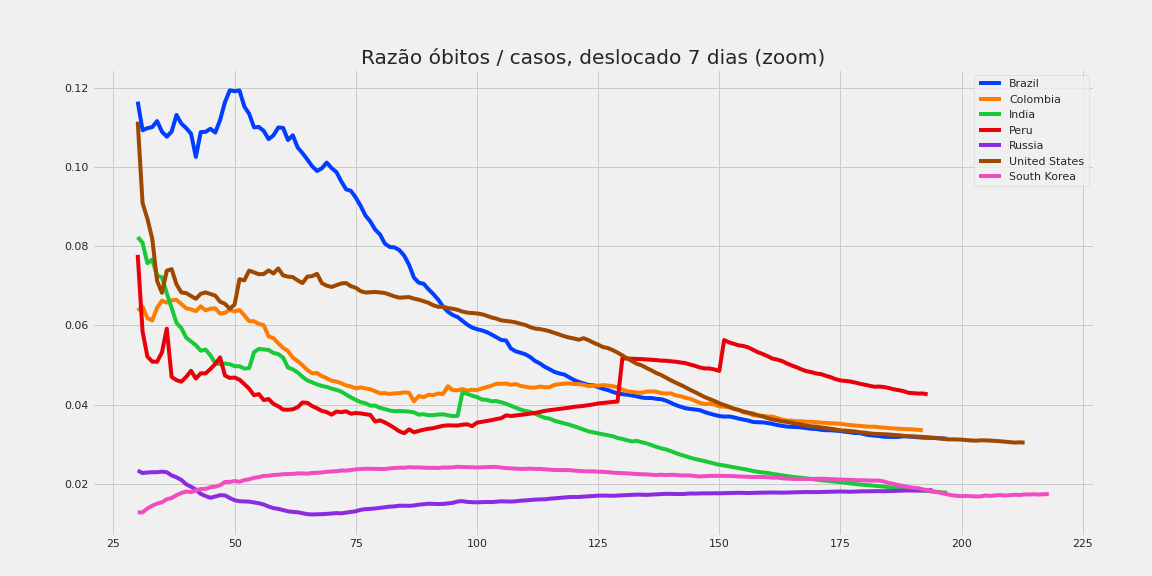 Exemplo de análise exploratória. Números baseados em dados públicos.
Exemplo de análise exploratória. Números baseados em dados públicos.
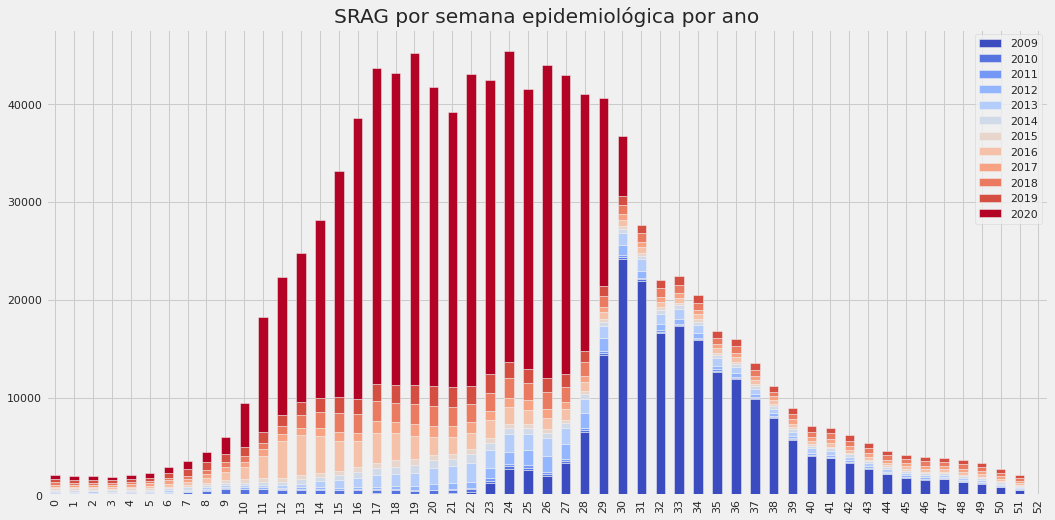 Exemplo de análise de Síndrome Respiratória Aguda Grave (SRAG). Números baseados em dados públicos.
Exemplo de análise de Síndrome Respiratória Aguda Grave (SRAG). Números baseados em dados públicos.
